Il Dna è un doppio filamento costituito da due catene di nucleotidi. Ciascun nucleotide è costituito da uno zucchero, il desossiribosio, un gruppo fosfato e una base azotata. Lo zucchero costituisce lo scheletro del singolo filamento e si trova all’esterno della struttura. Il legame che si instaura tra i diversi nucleotidi della singola catena è di tipo fosfodiesterico, il gruppo OH in posizione 3 del desossiribosio si lega al gruppo fosfato in posizione 5. Si usa quindi definire una estremità 3’ del filamento, laddove troviamo il gruppo in posizione 3 libero, e un’estremità 5’.
All’interno della struttura sporgono dai due filamenti le basi azotate. Queste basi possono essere quattro: adenina (A), timida (T), citosina (C) e guanina (G). Se consideriamo il singolo filamento, non esistono restrizioni nella sequenza con cui queste basi si susseguono. Se consideriamo invece la coppia di catene, riscontriamo un appaiamento obbligato tra le basi che si fronteggiano e cioè AT e CG. Ciò è dovuto al numero di legami che si formano tra le basi e alla distanza che le coppie occupano.
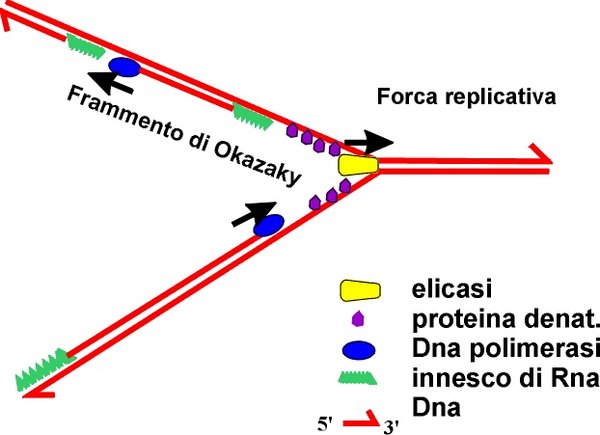
Durante la duplicazione del DNA, un enzima specifico, la DNA polimerasi, costruisce una nuova catena di DNA su ognuna delle due catene preesistenti, seguendo ovviamente la regola dell’appaiamento tra le basi. La costruzione del nuovo filamento avviene solo in direzione 5’->3’ per le caratteristiche intrinseche al legame stesso. Bisogna inoltre ricordare che questo enzima necessita di un innesco a RNA, cioè di una breve sequenza di RNA (RNA primer) da cui partire per sintetizzare il nuovo filamento, dal momento che è in grado solo di aggiungere nucleotidi a una catena preesistente.
Questa premessa è necessaria per spiegare i due tipi di attività esonucleasica che troviamo associati alla DNA polimerasi: un’attività esonucleasica 3’->5’ e una 5’->3’. L’attività esonucleasica è a carico di un sito della polimerasi stessa, distinto dal sito di polimerizzazione, o di una proteina del complesso. Esiste infatti più di una DNA polimerasi con caratteristiche distinte, sia in procarioti, quali Escherichia coli, sia negli eucarioti. Queste isoforme possono o no presentare attività esonucleasica.
Possiamo utilizzare come esempio la DNA polimerasi I di E. coli, che presenta entrambe le attività esonucleasiche. L’attività esonucleasica permette di excidere il nucleotide terminale della catena sia dall’estremità 3’ (3’->5’) che dalla 5’ (5’->3’). L’esonucleasi 3’->5’ permette alla DNA polimerasi di fare un passo indietro. L’azione è simile a quella che si fa durante la battitura di un testo con il tasto “delete”. Utilizzando lo stesso paragone, l’esonucleasi 5’->3’ equivale invece al tasto “cancel”. L’attività esonucleasica 3’->5’è la prima linea di difesa per correggere gli errori compiuti in fase di copiatura dalla DNA polimerasi stessa. Il tasso di incorporazione di nucleotidi sbagliati, che presentano quindi un errato appaiamento con il nucleotide corrispondente dell’altra catena, della polimerasi è infatti troppo elevato (circa 1 su 106). In un mammifero sono presenti circa 3X10 9 nucleotidi e quindi senza controllo il tasso di mutazione sarebbe altissimo (la mutazione è, in parole povere, proprio il cambiamento di uno o più nucleotidi nella sequenza originaria del DNA). La DNA polimerasi 3 riesce a riconoscere un appaiamento errato, forse grazie alle tensioni che si originano nella struttura, ferma l’avanzamento della polimerizzazione e torna indietro, excidendo i nucleotidi fino a quello appaiato scorrettamente. A questo punto riprende la polimerizzazione. Una polimerasi che presenti attività esonucleasica 3’->5’ viene definita “proof-reading”. L’importanza di tale catalisi enzimatica è tale che una mutazione che la riduca o la annulli è pressoché incompatibile con la vita.
Le cause dell’errato appaiamento delle basi sono varie e legate sia l’attività intrinseca della DNA polimerasi, sia alla possibilità delle basi di trovarsi transientemente in una forma tautomerica (detta rara ma che si presenta 1 volta ogni 104 -105 basi) che permette appaiamenti diversi dai canonici, sia da agenti esterni che vanno a ledere la struttura del DNA alterandolo chimicamente (alchilazione, ossidazione, etc.)
L’attività esonucleasica 5’->3’ è invece necessaria per eliminare i primer di RNA utilizzati come innesco della polimerasi, che vengono sostituiti da DNA. Ricordiamo che negli eucarioti sono presenti molteplici punti di innesco e quindi numerosi tratti di RNA che interrompono la catena di DNA neoformata. Inoltre, dal momento che l’attività polimerasica avanza solo in direzione 5’->3’, un filamento viene duplicato in maniera continua mentre l’altro viene duplicato in frammenti, con numerosissimi inneschi di RNA. Su quest’ultimo filamento l’attività esonucleasica 5’->3’ è quindi indispensabile.
Per un riassunto delle possibili mutazioni nel DNA si veda:
http://www.unicz.it/didattica/lauree/biotecnologie/dispense/biologiamolecolare/cuda/RiparazioneDNA.pdf
Per ulteriori dettagli sulla duplicazione del DNA, la cui spiegazione qui sarebbe troppo complessa, si vedano: www.unicz.it/didattica/lauree/biotecnologie/dispense/biologiamolecolare/cuda/ReplicazioneDNA.pdf it.wikipedia.org/wiki.cgi?Dna