Le tecniche di microarray si basano sul principio della microibridazione contemporanea di migliaia di frammenti di DNA con specifiche sonde o "probe" fissate su un supporto solido (chip). Il chip è generalmente una matrice di plastica, vetro o silicio trattata chimicamente per favorire l’adesione delle sonde al supporto ed è costituito da vari spot che contengono ognuno numerose copie di una sonda complementare ad un gene o ad una particolare sequenza nucleotidica di interesse.
La tecnica, nata negli anni ’90, si è poi diversificata in più varianti a seconda delle applicazioni e del tipo di campione da esaminare (per analisi di espressione genica o per il sequenziamento e ricerca di SNPs) o in base alla modalità di costruzione delle sonde, per cui si distinguono le metodiche di microarray spottati e microarray di oligonucleotidi ad alta densità.
Nel caso dei microarray spottati, le sonde sono oligonucleotidi o piccoli frammenti di cDNA prodotti mediante PCR e successivamente stampati in modo sequenziale su vetrino negli specifici spot, mediante un passaggio UV-crosslinking che ne favorisce la fissazione alle cariche positive del vetrino stesso.
Generalmente questo tipo di tecnica è impiegata nell’analisi in simultanea di due cDNA distinti (es. campione e controllo), marcati con due diverse molecole fluorescenti, il ché permette di miscelarli e ibridizzarli in contemporanea sfruttando la competizione di legame tra i due cDNA target con la probe. Attraverso uno scanner è possibile registrare la fluorescenza emessa in corrispondenza di ogni singolo spot, in questo modo si possono ottenere tre diverse combinazioni: nel caso nessuno dei due campioni abbia affinità per la sonda in esame non verrà registrato alcun segnale di fluorescenza, se solo uno dei campioni si legherà alla sonda verrà registrata la fluorescenza del colore corrispondente al campione legato, in cui l’intensità del segnale è direttamente proporzionale al numero di fluorofori associati, se entrambe le sonde hanno sequenze complementari alla probe si genera invece un segnale di colore intermedio.
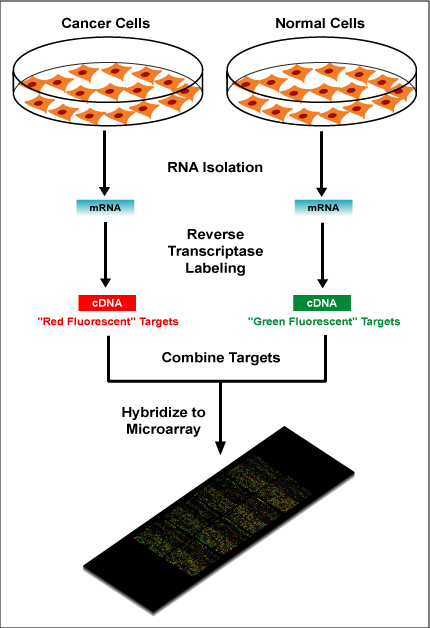
Esemplificazione della tecnica di microarray spottato attraverso cui è possibile esaminare i profili di espressione di due diversi campioni cellulari (wikipedia: http://it.wikipedia.org/wiki/
Nei microarray di oligonucleotidi ad alta densità, gli oligonucleotidi che fungono da sonda sono sintetizzati in situ attraverso un procedimento di fotolitografia. In dettaglio, il processo prevede la presenza di specifici linker su vetrino che si trovano in forma protetta, la loro attivazione avviene mediante esposizione luce diretta che smaschera i gruppi protettivi dei linker per incorporare il primo nucleotide della probe. Il processo di polimerizzazione segue lo stesso principio luce-dipendente con la rimozione dei gruppi protettivi associati ai nucleotidi; attraverso particolari maschere che agiscono da filtro è possibile procedere in modo selettivo nell’attivazione dei diversi spot, generando nel complesso il proprio assetto di sonde che caratterizza la matrice di ibridazione.
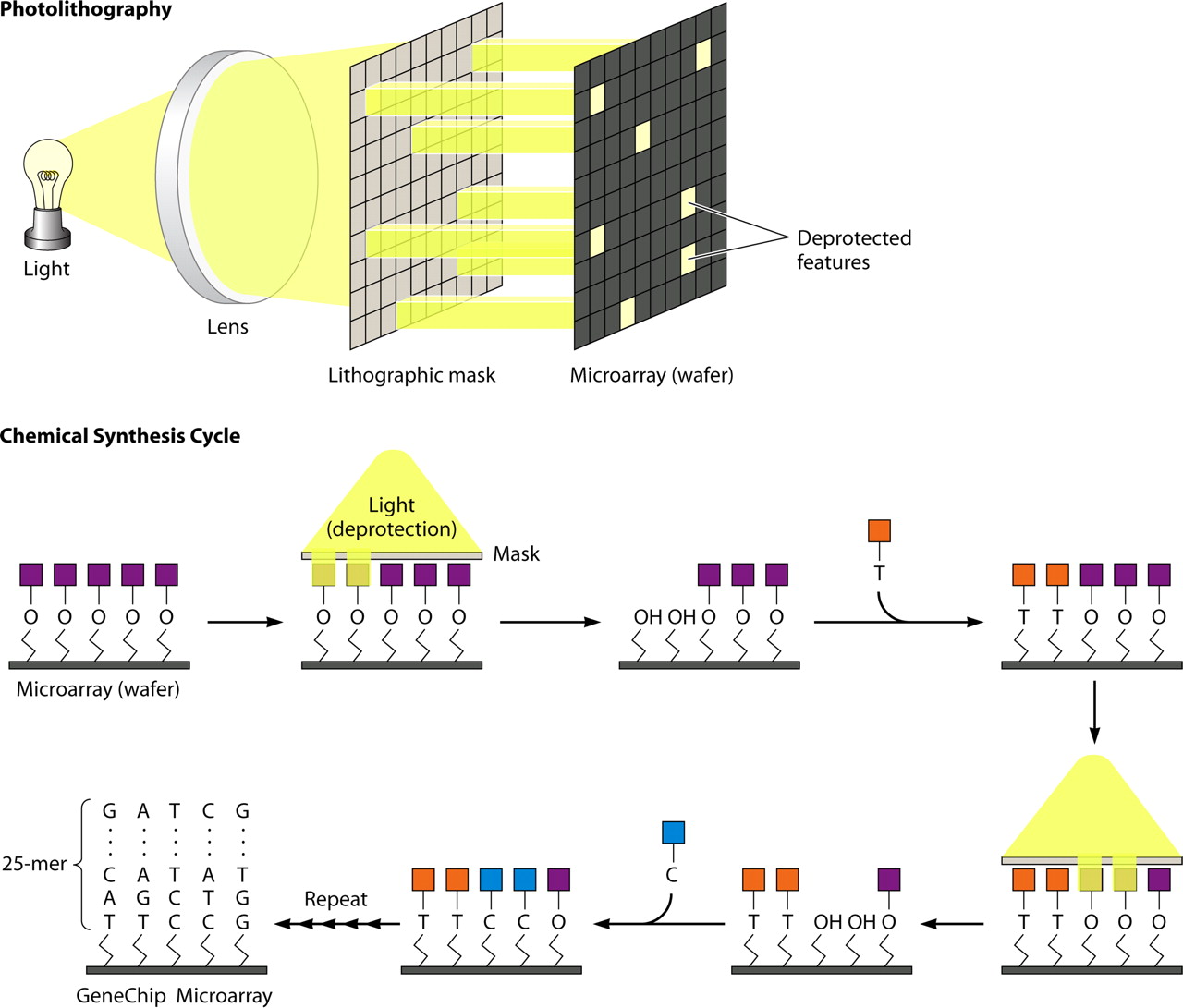
Passaggi per la costruzione delle sonde ad oligonucleotidi tramite processo fotolitografico (Clin. Microbiol. Rev. October 2009 vol. 22 no. 4 611-633 "Basic Concepts of Microarrays and Potential Applications in Clinical Microbiology")
Al contrario dei microarray spottati, quelli ad oligonucleotidi ad alta densità consentono l’analisi di un singolo campione su ogni gene chip, la successiva scannerizzazione permette di stabilire il grado di ibridizzazione tra il DNA in analisi, generalmente marcato con il sistema fluorescente biotina-streptavidina/ficoeritrina, con le diverse sonde create.
Nel complesso le due metodiche presentano diversi campi di impiego: i microarray spottati trovano maggiori applicazioni nel confronto dell’espressione genica tra una linea di studio e una linea di controllo, ad esempio per studiare il profilo di espressione tra cellule tumorali e cellule normali oppure nella comparazione di DNA tra pazienti e persone sane. Di contro i micorarray ad alta densità consentono la costruzioni di array ad alto contenuto di informazioni (un singolo array di 1,28 cm² contiene un probe set per 40.000 geni) al fine di riconoscere in modo selettivo particolari sequenze o scannerizzare grosse regioni genomiche di vari organismi viventi.