Il lateral flow, western blotting, micro e macro array sono tecniche rappresentative di tre diversi principi analitici:
1 – Tecniche immunochimiche, sono quelle che utilizzano reazioni tipiche del sistema immunitario come la relazione tra antigene e anticorpo per determinare la presenza di componenti o contaminanti di piccole o grandi dimensioni quali antimicrobici, micotossine, droghe, pesticidi, anabolizzanti, ormoni, allergeni, OGM, ecc. Si utilizzano sia per diagnostica umana, animale in campo ambientale o in agricoltura per la determinazione di analiti. L’elevata specificità degli anticorpi permette di rilevare quantità di antigene in matrici complesse e molto ridotte. I test immunochimici sono rapidi, stabili nel tempo, non richiedono strumentazione per cui sono abbastanza economici e la loro semplicità d’uso li rende eseguibili da personale senza particolari qualifiche anche sul campo.
Gli anticorpi (o immunoglobuline) sono proteine presenti nel sangue che hanno il compito di riconoscere e legarsi specificatamente ad antigeni presenti sulla superficie di corpi estranei (virus, batteri, tossine, ecc.). Il tipo di legame è altamente specifico e simile a quello che c’è fra una chiave e la propria serratura.
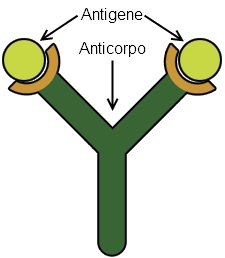
Tra le tecniche immunochimiche più diffuse abbiamo l’Elisa, i test di agglutinazione, l’immunodiffusione e il Lateral flow.
Il lateral flow è un test qualitativo (presenza/assenza), in alcuni casi semi-quantitativo, di cui l’esempio più noto è il test di gravidanza disponibile in farmacia. Il supporto, generalmente di forma rettangolare e allungata, è composto da strati sovrapposti di materiale poroso capace di trasportare liquidi per capillarità da un’estremità all’altra. Genericamente il primo strato agisce come una spugna che assorbe il campione liquido e lo trasferisce allo strato sottostante sul quale si trovano anticorpi (chiamiamoli anticorpi tipo 1) coniugati a molecole indicatrici (in genere si tratta di un indicatore colorato). Se il campione in esame possiede l’antigene ricercato questo si lega agli anticorpi coniugati e inizia a migrare passivamente verso il lato opposto del supporto. Durante la migrazione il complesso incontra, in una determinata posizione, una seconda serie di anticorpi (anticorpi tipo 2) specifici per lo stesso antigene bersaglio, ma fissati al supporto i quali catturano il complesso migrante antigene/anticorpo1/indicatore e lo bloccano in sito. La concentrazione in un punto degli anticorpi coniugati rende l’indicatore riconoscibile a vista come una banda colorata.

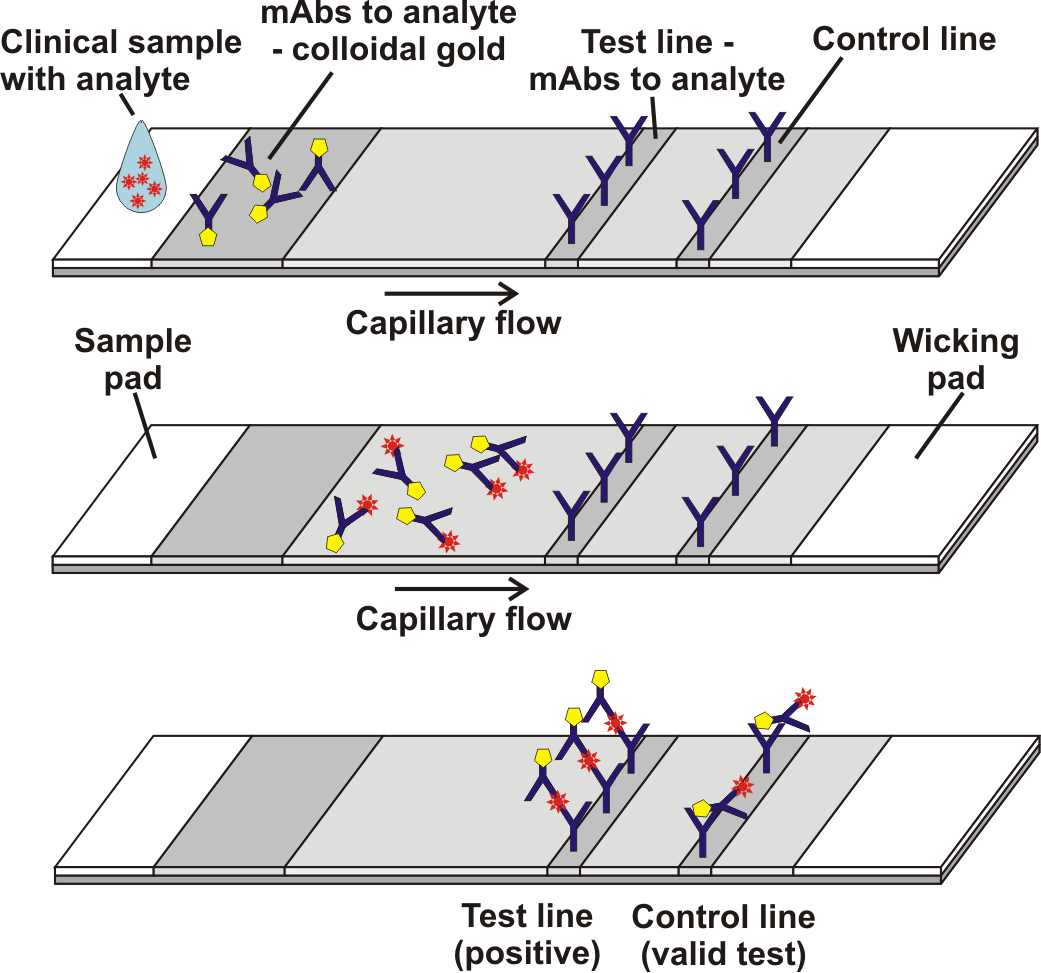
Questo tipo di test è chiamato non competitivo o diretto, ma esistono anche test lateral flow di tipo competitivo in cui il complesso migrante antigene/anticorpo1/indicatore non si lega agli anticorpi fissati al supporto perché non ha siti di legame liberi e si lega solo alla linea di controllo.
2 – Tecniche elettroforetiche, consistono nella separazione di molecole elettricamente cariche tramite l’applicazione di un campo elettrico. Queste tecniche sono usate per separare proteine, acidi nucleici o altre piccole molecole.
Come funziona la separazione di molecole cariche?
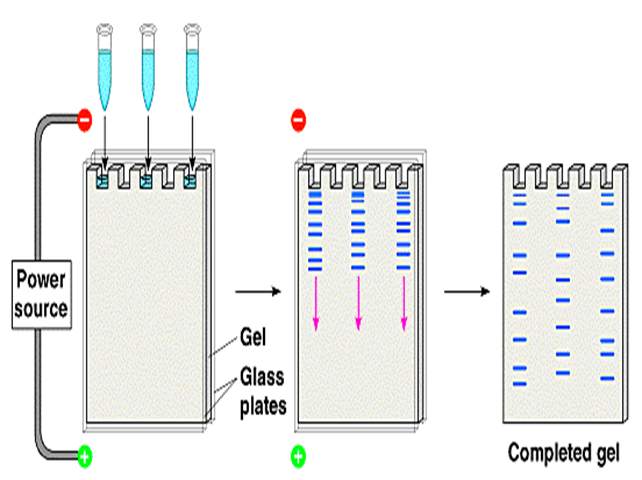
La velocità di una molecola carica che si muove in un campo elettrico è direttamente proporzionale alla forza del campo elettrico ed alla carica della molecola ed è inversamente proporzionale alle dimensioni della molecola ed alla viscosità del mezzo in cui si muove. L’elettroforesi è un metodo che permette di separare le molecole in base alle loro dimensioni, alla loro carica elettrica e alla forma.
Ipotizziamo di dover separare dei frammenti di DNA che sono molecole con carica negativa. Inseriamo i campioni di DNA in corrispondenza del polo negativo di un foglio di gel attraverso cui le molecole dovranno passare. All’accensione del campo elettrico i frammenti di DNA inizieranno ad essere attratti e quindi a muoversi verso il polo positivo posto all’altra estremità. Nella loro migrazione i frammenti più piccoli riusciranno a passare meglio nel reticolo del gel e saranno di conseguenza più veloci dei frammenti grandi. Trascorso un certo tempo, il campo elettrico viene interrotto e si potranno osservare nel gel delle bande dovute alla concentrazione di frammenti di DNA della stessa dimensione che hanno viaggiato alla stessa velocità. I frammenti che hanno percorso più distanza corrispondono a quelli di minori dimensioni, andando verso il polo negativo troveremo bande con frammenti via via più grandi.
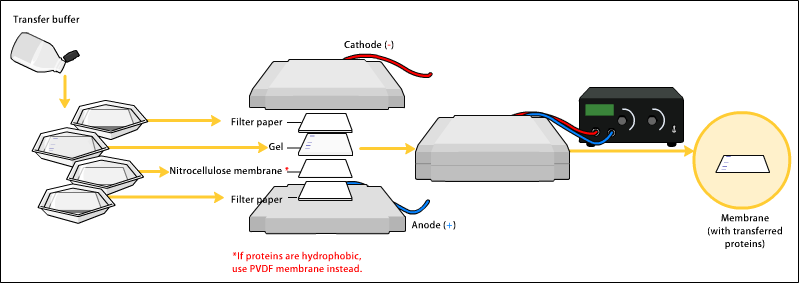
Il western blot viene utilizzato per determinare la presenza e la quantità di una specifica proteina ed è la risultante dell’accoppiamento di una tecnica immunochimica con una tecnica elettroforetica. Le proteine vengono inizialmente separate per elettroforesi, quindi vengono trasferite su una membrana di nitrocellulosa o di polivinildenfluoruro (PVDF) in grado di legare i polipeptidi. Si crea un “sandwich” in cui il gel dell’elettroforesi con la membrana di trasferimento vengono posti all’interno di strati di carta da filtro e spugna. Il “sandwich” viene inserito tra due elettrodi a piastra immersi in un tampone elettrolitico ai quali viene applicata una differenza di potenziale che provoca la migrazione delle proteine dal gel di elettroforesi alla membrana mantenendone la posizione. Dopo il trasferimento la membrana viene fatta incubare con anticorpi specifici per la proteina ricercata coniugati ad una molecola rilevatrice (sonda) che in seguito verrà evidenziata tramite colorazione, chemioluminescenza o rilevatori di fluorescenza.
3 – Tecniche di analisi degli acidi nucleici, vengono utilizzate nei campi più disparati per l’identificazione di specie, caratterizzazione di alcuni componenti, tracciabilità e indagini microbiologiche; includono metodi rapidi con elevata specificità e sensibilità.
Gli acidi nucleici sono molecole presenti nelle cellule e deputate al trasporto e alla conservazione dell’informazione genetica, ne fanno parte il DNA (acido desossiribonucleico) e l’RNA (acido ribonucleico). Tutti gli organismi viventi hanno acidi nucleici, solitamente entrambi, ma a volte anche solo uno dei due come nei virus.
Il DNA è composto da due filamenti avvolti uno sull’altro a formare una doppia elica. Ogni filamento è a sua volta costituito da una serie di 4 basi azotate (adenina, guanina, citosina, timina) che si legano con quelle dell’altro filamento in modo complementare unendo le catene. La guanina si lega solo alla citosina e la timina solo all’adenina. L’RNA ha la stessa struttura, ma manca la timina che viene sostituita dall’uracile ed è a singolo filamento.
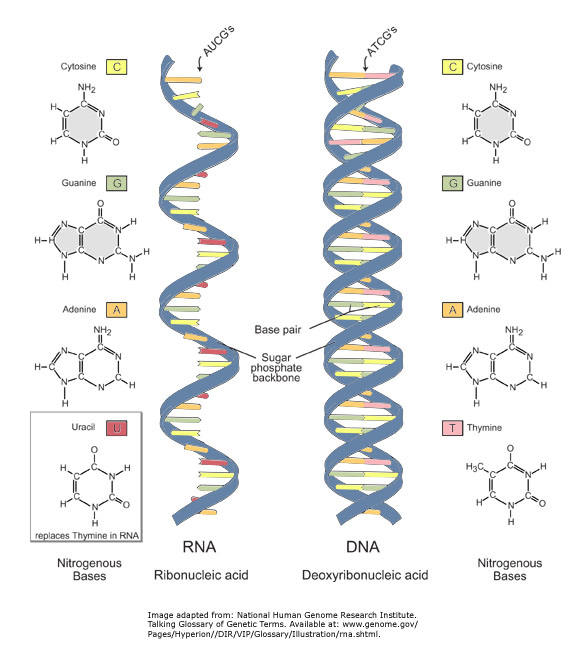
DNA –> mRNA –> proteine, peptidi
I DNA microarrays anche noti come DNA chip o chip genomici, sono una moderna tecnica di indagine biomolecolare che permette di analizzare contemporaneamente un gran numero di geni sfruttando le capacità di ibridizzazione del DNA singolo con il suo filamento complementare. Il DNA microarray consiste in un supporto di vetro, plastica o silicone composto da pozzetti all’interno dei quali vengono fissati alla superficie con legami covalenti o non covalenti dei filamenti singoli di DNA corrispondenti a parti di geni specifici noti (sonde).
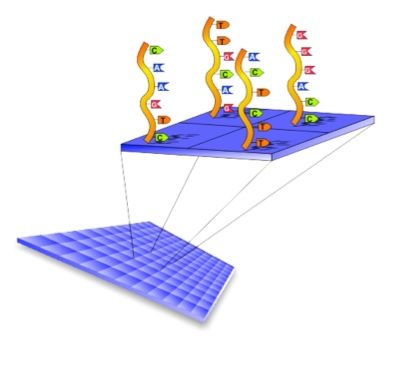
Gli array si basano sul principio che due filamenti di DNA a singola elica possono rinaturarsi quando riconoscono regioni complementari alla loro, formando molecole ibride.
Nello specifico, l’RNA prelevato dal tessuto da esaminare viene retrotrascritto (trascrittasi inversa) in cDNA (DNA complementare) con l’uso di nucleotidi1 fluorescenti ed eventualmente amplificato con la PCR (polymerase chain reaction) . Il cDNA marcato ottenuto viene messo su piastra in modo da accoppiarsi con i filamenti di cDNA di geni noti posti nei pozzetti. Quindi si procede al lavaggio per eliminare il cDNA non legato e si analizza la fluorescenza per evidenziare su quali pozzetti è avvenuto il legame. Utilizzando dei sistemi di lettura automatizzati è possibile anche quantificare l’intensità della fluorescenza per avere una stima della concentrazione di cDNA e quindi di RNA nel campione in esame.
.png)
Gli array vengono utilizzati per l’analisi dell’espressione genica, per la genotipizzazione (un chip può contenere anche sequenze geniche di specie diverse per effettuare un’analisi simultanea), oppure per l’analisi dell’antibiotico resistenza.
I macroarray sono solamente microarray più grandi, si tratta di termini intercambiabili che si usano per differenziare il numero di pozzetti per chip o le dimensioni degli stessi.
____
1 il nucleotide è l’unità molecolare dell’acido nucleico. Ogni nucleotide è composto da una base azotata, uno zucchero e un gruppo fosfato