La Variegazione per Effetto di Posizione (Position Effect Variegation o PEV) è un silenziamento genico che si manifesta con un fenotipo a mosaico, dove ciascun gruppo di cellule clonali presenta l’inattivazione o l’attivazione del gene interessato da PEV in maniera apparentemente casuale.
A livello genico, la PEV è causata dallo spostamento, solitamente per inversione o per effetto di trasposoni, di un determinato gene in vicinanza di una regione eterocromatinica. Nella PEV il silenziamento genico viene ereditato in maniera epigenetica nelle cellule figlie, cioè senza un cambiamento nella sequenza nucleotidica. Nel caso di silenziamento epigenetico, un gene inattivo o attivo in una cellula, rimarrà tale probabilmente anche nelle cellule figlie e così via, fino a che non intervenga un qualche fattore che ne modifichi lo stato.
Per comprendere i meccanismi della PEV dobbiamo ricordare che negli eucarioti superiori la cromatina nel nucleo si presenta sotto due forme facilmente distinguibili: eucromatina, decondensata e trascrizionalmente attiva, ed eterocromatina, condensata e trascrizionalmente inattiva.
L’eterocromatina differisce dall’eucromatina sia per l’aspetto citologico che per l’organizzazione delle sequenze. L’eterocromatina appare relativamente condensata durante l’interfase, si colora differentemente dall’eucromatina in metafase e partecipa alle associazioni non omologhe. Le sequenze che si trovano nell’eterocromatina sono costituite tipicamente da blocchi di DNA altamente e mediamente ripetuto contenente pochi geni.
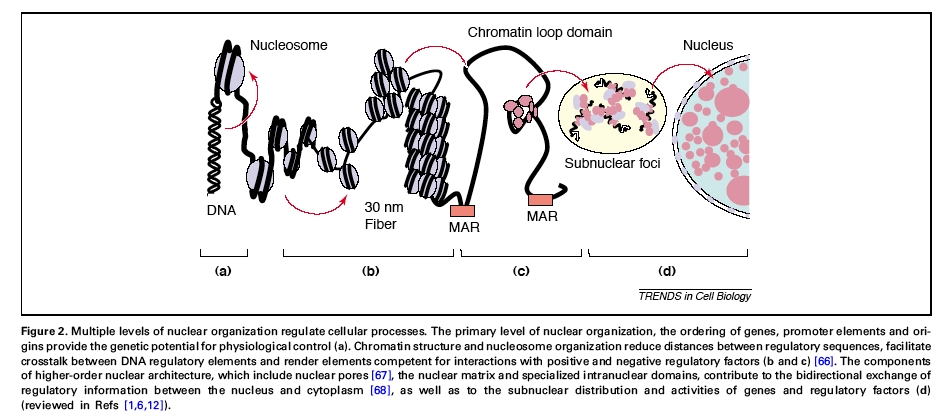
Quando i geni dell’eucromatina si muovono in vicinanza dell’eterocromatina, mostrano un’espressione a mosaico, cioè la PEV. La PEV è stata osservata per la prima volta da Muller nel 1930 in Drosophila, nella quale alcuni riarrangiamenti genici causavano la repressione di un certo numero di geni, muovendoli in vicinanza di un grande blocco di eterocromatina centrale. In particolare la variegazione si esprimeva fenotipicamene con un occhio a chiazze bianco/rosse quando era interessato da questo spostamento il gene white. La prima osservazione, utile per la comprensione dei meccanismi implicati nell’espressione variegata di white, fu che l’entità della repressione differiva in base alla vicinanza del gene all’eterocromatina
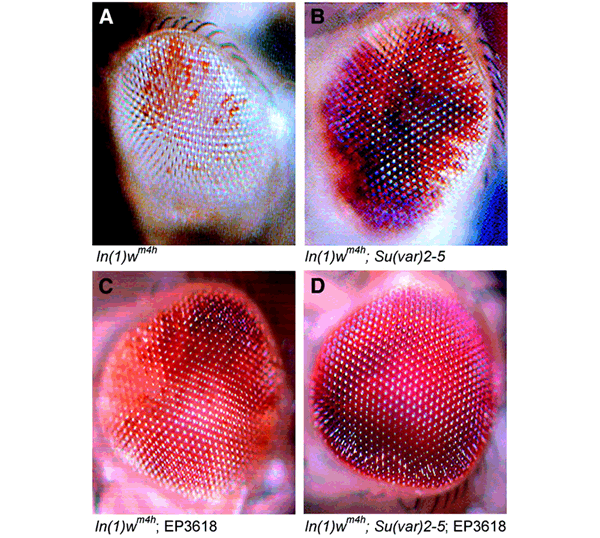
PEV in Drosophila
EMBO reports 2, 3, 211–216 (2001)
doi:10.1093/embo-reports/kve09
Published online: March 2001
. Inversioni che portavano white più vicino all’eterocromatina, causavano una repressione maggiore.
Nella PEV l’eterocromatina sembra diffondersi nell’eucromatina in maniera polare, infatti i loci contenenti geni interessati da questo tipo di silenziamento presentano caratteristiche eterocromatiniche.
Un’interessante ed esplicativa osservazione è che la PEV viene soppressa dall’aggiunta di regioni eterocromatiniche mentre viene incrementata dalla rimozione delle stesse. Questo fenomeno può essere spiegato assumento che l’eterocromatina aggiunta competa con l’eterocomatina endogena per il reclutamento dei fattori eterocromatinizzanti.
Una seconda domanda alla quale si è data una risposta è se l’eterocromatinizzazione del DNA fosse dovuta alla presenza di sequenze specifiche o al solo fatto che queste sequenze fossero altamente ripetute. Inserendo una serie di sequenze più o meno ripetute del gene white in Drosophila, ottenute come duplicazioni strettamente collegate a un trasposone, che porta un mini gene white, e studiando i gradi di PEV ottenuti, si è dimostrato che l’eterocromatina si forma a causa della natura ripetuta delle sequenze che ne fanno parte e non a causa della presenza di sequenze specifiche.
Drosophila ad esempio, non avviene la metilazione del DNA, quindi le speculazioni sul marcatore per il mantenimento dello stato si sono focalizzate su una particolare conformazione comatinica o su un proteina che leghi il DNA in grado di persistere attraverso la fase S e la mitosi.In Drosophila, la proteina GAGA, un regolatore trascrizionale codificato da un gene E(VAR), si lega a molti siti nell’eucromatina e a specifici blocchi nell’eterocromatina. GAGA rimane legata a una sequenza satellite ricca di A-G durante tutto il ciclo cellulare. Tuttavia in alcuni tipi cellulari, quali i neuroblastomi larvali, si lega all’eterocromatina solo durante l amitosi. Nell’ebrione tardivo, frazioni significative di HP1 e SU(VAR)3-7 disperdono dai cromosomi durante la metafase e quindi si riassemblano nella cromatina alla telofase.
Questi complessi schemi di dispersione delle proteine e loro riassemblamento nella cromatina a diversi momenti del ciclo cellulare, hanno implicazioni nella PEV e nell’espressione genica in generale, dal momento che definiscono una finestra di opportunità per il rimodellamento dxella cromatina.
Mentre i fattori di trascrizione vengono spostati dal promotore durante la metafase, rimane un’impronta sul DNA e la persistenza di questo marcatore, non ancora identificato, sul cromosoma potrebbe spiegare il ristabilirsi dello stato trascrizionale dopo la mitosi. Un tipo simile di “segnalibro” molecolare potrebbe spiegare come viene ereditato lo stato attivo o inattivo dai geni che presentano PEV. 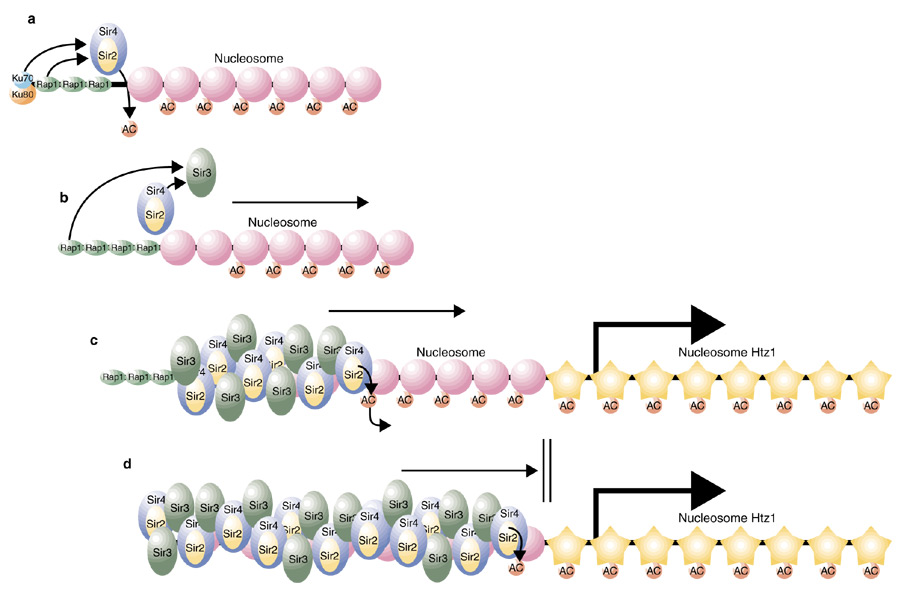
Azione dei fattori eterocromatinizzanti (SIR) nel lievito e dei fattori terminatori della eterocromatina (nucleosomi contenti Hitz).
Nature Cell Biology 5, 278 – 280 (2003)
doi:10.1038/ncb0403-278
Per una spiegazione su eterocromatina ed eucromatina:
Differenza tra eu ed eterocromatina
Alcuni riferimenti a geni implicati nella formazione dell’eterocromatina in diverse specie animali
link 1