Il periodo preimpianto dell’embrione di mammifero inizia dalla fecondazione (stadio di zigote) e termina con l’impianto nella parete uterina, cioè con la formazione di quelle strutture che andranno a costituire la placenta. Questo avviene poco dopo la formazione della blastocisti.
Inizialmente l’embrione subisce una segmentazione che porta il numero di cellule da 1 a 16-32. Successivamente si ha un processo chiamato compattazione dal quale si origina la morula.
Infine nella morula si differenziano due popolazioni cellulari, il trofoblasto e la massa cellulare interna (ICM). Ques’ultima darà origine all’embrione e a pochi annessi embrionali quali il sacco vitellino. Il trofoblasto invece prenderà parte alla formazione della placenta.
Il periodo preimpianto porta a una modificazione profonda dell’embrione ed è un periodo di attiva sintesi proteica.
Alcune proteine, anche regolatrici, sono di eredità materna, cioè si trovano già nella cellula uovo sotto forma o di mRNA impacchettati e pronti per la traduzione successiva, o di proteine. Un esempio è il gene MATER, implicato in maniera cruciale nei primissimi stadi della segmentazione. I topi mancanti del gene o che ne presentano una copia difettosa sono sterili o producono embrioni che si bloccano allo stadio di due cellule.
Nell’elenco dei geni materni possiamo citare anche Hsf1, le proteine della zona pellucida e le E-caderine, queste ultime implicate nei processi di adesione cellulare.
Moltissime proteine devono invece essere create ex-novo dall’embrione, le cui cellule dovranno conseguentemente determinare quali geni attivare e in che successione spazio-temporale. I prodotti dei geni trascritti possono essere proteine strutturali, cioè implicate nella costruzione della cellula (tubulina, actina, ecc.), enzimi o fattori di regolazione della trascrizione (ricordiamo tra gli homeobox il gene Oct4, implicato nel mantenimento dello stato indifferenziato delle cellule).
Gli studi effettuati sull’embrione preimpianto hanno preso in considerazione soprattutto il topo, il ratto e il coniglio. Alcuni dati sono stati ottenuti anche in specie di interesse zootecnico allo scopo di migliorare le tecniche di fecondazione artificiale. Questi ultimi, ad esempio, presentano un periodo preimpianto dilatato, con uno stadio di blastocisti molto più lungo.
A parte queste piccole differenze, i meccanismi alla base dello sviluppo precoce dell’embrione di mammifero sono simili in tutti i mammiferi studiati.
Una spinta importante per la comprensione dei meccanismi di attivazione genica nell’embrione precoce è stata fornita dagli studi sulle cellule staminali embrionali (ESC). I risultati di questi studi comunque sono stati ottenuti in vitro e ancora pochi sono stati confermati da esperimenti in vivo.
Pre quanto riguarda i geni più precoci, allo stadio di zigote esiste una finestra temporale definita zigotic genome activation (ZGA, attivazione genomica dello zigote), che segna l’inizio della trascrizione dei geni zigotici. Tra questi possiamo citare come esempio U2afbp-rs, hsp70.1, mTEAD-2, eIF-4C e MuERV-L. Per la maggior parte di questi geni le funzioni e i meccanismi di azione sono ancora controversi. MuERV-L, ad esempio è coinvolto nella formazione della placenta e nella temporizzazione degli eventi zigotici, ma non se ne conosce ancora il preciso pattern di azione.
Sempre a livello del periodo di ZGA vengono attivati e trascritti i geni per molte ATPasi, quali la 6, componenti del citoscheletro, actina e tubulina.
Qui di seguito è riportato un elenco di geni attivi identificati in embrioni bovini nel periodo preimpianto :
– 60S Ribosomal protein L7
– Proteasome 26S subunit ATPase (PSMC3)
– Phosphoglycerate kinase 1
– 40S Ribosomal protein S25
– Ribosomal protein L27
– Chaperonin 10
– SH3-domain protein 5 (Ponsin)
– Glycine receptor b subunit
– Ribosomal protein L18a
– Activated RNA polymerase II transcripton cofactor 4
– Casein kinease II a subunit
– Cytochrome C oxidase subunit VII
– Tropomyosine
– Mannosidase a Class 2A member 1
– Transcription elongation factor B
– Mitochondrial ribosomal protein
– Chloride intracellular channel
– Eukaryotic translation initiation factor (EIF5)
– Zinc finger protein 277 (2NF 277)
– Opa-interacting protein OIP2
– RNA polymerase I (DNA directed)
– Nucleosome assembly protein 1-like (NAP ILI)
– Mitochondrial ribosomal protein (MRP L24)
– Ribosomal protein S26
– DC 13 protein
– Deoxyguanosine kinase
– Mitochondrial ribosomal protein L20
– Mariners transposase gene
– Mitochondrial ATP synthesis d subunit
Una trattazione completa dell’espressione zigotica dei mammiferi preimpianto implicherebbe una monografia molto lunga e complessa. Consiglio una ricerca settoriale sui singoli aspetti dello sviluppo.
Attraverso questo link è possibile accedere ai principali database per lo sviluppo del topo
http://www.rodentia.com/wmc/domain_genome.html
In questo sito è possibile ottenere ulteriori informazioni anche su altri mammiferi.
http://www.ucalgary.ca/UofC/eduweb/virtualembryo/
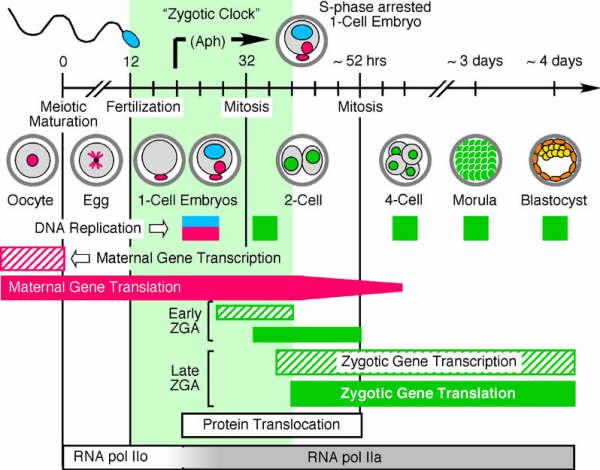
Fig 1
Transizione da geni materni a geni zigotici nell’embrione precoce del topo. In rosso sono indicati gli eventi materni, in blu quelli paterni e in verdi i meccanismi zigotici.
In giallo è indicata la massa cellulare interna, in arancione il trofoectoderma.
Le barre tratteggiate indicano i periodi di trascrizione dei geni, le barre solide indicano i periodi di traduzione dei geni.