Questo tipo di analisi viene eseguito utilizzando tecniche di citogenetica molecolare. La citogenetica studia il corredo cromosomico cellulare e le sue eventuali alterazioni. Il termine “molecolare” si riferisce alla possibilità di analizzare la struttura cromosomica fin nei suoi più minuti dettagli, cioè fino alle singole molecole costituenti. Le indagini di tipo citogenetico sono normalmente utilizzate per la diagnosi prenatale e postnatale delle malattie congenite e per l’identificazione delle anomalie cromosomiche tipiche di alcuni tumori.
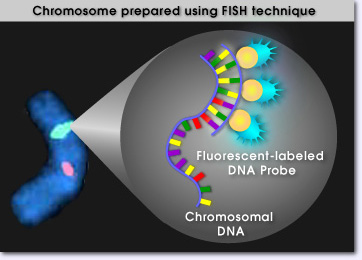
La specifica localizzazione di un gene sul cromosoma può essere determinata grazie ad una indagine citogenetica nota come ibridazione a fluorescenza in situ, spesso indicata con l’acronimo inglese FISH (Fluorescence In Situ Hybridization). Questa tecnica si basa sulla disponibilità in laboratorio di una sonda, cioè di una copia complementare del gene da localizzare, marcata con una sostanza fluorescente. Messa in contatto col corredo cromosomico cellulare, la sonda ibridizza con il suo gene, cioè lo riconosce e vi si lega in modo specifico, un po’ come fanno due pezzi complementari di un puzzle. Il complesso sonda-gene viene poi visualizzato tramite microscopia a fluorescenza.
Esistono diverse decine di protocolli di laboratorio per la FISH, ciascuno dei quali è stato elaborato specificatamente in base al gene da ricercare e al campione cellulare di partenza. Non potendo entrare nello specifico di ognuno di essi, ci si accontenterà qui di descriverne le comuni fasi principali.
- Preparazione della sonda – La sonda può essere prodotta tramite amplificazione del gene in questione per PCR (Polymerase Chain Reaction) oppure derivare da un clone di una libreria genomica. Nel caso di sonde prodotte per PCR, la marcatura fluorescente viene ottenuta grazie all’incorporazione di nucleotidi coniugati (per esempio con fluoresceina isotiocianato, o FITC). Nel caso di sonde derivate da cloni genomici, i nucleotidi marcati vengono inseriti tramite una reazione enzimatica (nick translation) che sfrutta le proprietà della DNA polimerasi.
La marcatura della sonda può essere di tipo diretto (il nucleotide è legato direttamente alla sostanza fluorescente) oppure di tipo indiretto (il nucleotide è legato ad una molecola che viene poi riconosciuta dalla sostanza fluorescente, per esempio nucleotide-biotina riconosciuto da avidina-fluorocromo) . - Raccolta e coltura del tessuto campione – La FISH può essere eseguita su preparazioni cromosomiche o nuclei interfasici di cellule derivate da: biopsie tumorali o tessuti fetali abortivi, villi coriali, liquido amniotico, sangue periferico, midollare o funicolare.
Le condizioni di coltura variano a seconda della tipologia di campione. Il materiale bioptico, per esempio, viene frammentato meccanicamente con un bisturi o enzimaticamente con collagenasi. La sospensione cellulare viene lavata e quindi posta in adeguato terreno di coltura, in sospensione in fiasca oppure in situ su vetrino.
Il sangue prelevato, invece, viene trattato con 500 unità/ml di eparina (un anticoagulante) ed incubato a 37 °C in terreno di coltura RPMI. - Preparazione del campione – Le cellule contenute nel campione vengono trattate con un inibitore della mitosi (Colcemid 0.02 μg/ml per alcune ore) e quindi con una soluzione ipotonica (0.075 M potassio cloruro). Questi due trattamenti hanno lo scopo di bloccare il ciclo cellulare in metafase e facilitare la separazione e l’identificazione dei cromosomi al centro della cellula. Il preparato viene quindi fissato con metanolo ed acido acetico in proporzione 3:1 e montato su vetrino.
- Ibridazione e visualizzazione. – Sia la sonda sia il preparato cromosomico vengono riscaldati per denaturarne il DNA. La sonda viene quindi applicata al vetrino. L’ibridazione avviene a 37 °C per un periodo di tempo variabile fra 16 e 48 ore. Per rimuovere un eventuale eccesso di sonda e così ridurre una possibile ibridazione non specifica, il preparato viene lavato con formamide e una soluzione di sodio citrato e sodio cloruro (soluzione SSC). Nel caso di sonde a marcatura indiretta, per esempio con biotina, il complesso avidina-fluorocromo dovrà essere aggiunto al preparato prima dell’osservazione al microscopio a fluorescenza. Durante l’osservazione in microscopia, il punto del cromosoma ove la sonda avrà ibridato apparirà come un puntino fluorescente. Questo “puntino” corrisponde alla localizzazione sul cromosoma del gene analizzato.
I dati ricavati dal sequenziamento del genoma umano, ormai completato, e da quello del genoma di svariati organismi, tuttora in corso, ci permettono di conoscere esattamente da quali geni sia composto il corredo cromosomico di un organismo ed in che ordine tali geni siano disposti. Poiché queste informazioni sono disponibili per la consultazione in rete, è inoltre possibile, qualora lo scopo della ricerca non sia diagnostico ma puramente conoscitivo, localizzare “virtualmente” un gene sul rispettivo cromosoma con l’ausilio di soli strumenti informatici. Per chi sia interessato a questo tipo di approccio, ecco qui un breve esercizio guidato:
- andate sul sito http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?txid=9606 (si tratta della pagina del portale NCBI, National Center for Biotechnology Information, dedicata al genoma umano).
- inserite nel riquadro in alto a sinistra (vicino a “Search for”) il nome inglese del gene che si volete localizzare (per esempio, dystrophin, un gene implicato nella distrofia muscolare).
- cliccate su “Find”. Sullo schermo comparirà il corredo cromosomico umano con le posizioni dei geni della distrofina e utrofina (una proteina omologa alla distrofina) evidenziate in rosso
- cliccando su DMD o DRP2 o UTRN, si otterà un ingrandimento della mappa cromosomica attorno alla posizione del gene selezionato.
Provate a cercare i geni di vostro interesse oppure a curiosare su altre pagine di questo sito, che è uno dei più utilizzati da chi si occupa di genomica.
http://gslc.genetics.utah.edu/units/disorders/karyotype/images/FISH_technique.jpg