Dal punto di vista chimico le proteine sono di gran lunga le macromolecole più complesse come struttura e funzione, questa complessità è il risultato di miliardi di anni di storia evolutiva che ne ha plasmato sia la morfologia che l’attività.
Le proteine sono catene lineari di aminoacidi legati da particolari legami covalenti detti “peptidici”; ogni aminoacido possiede la propria catena laterale che ne conferisce specifiche proprietà.
In cellula esistono 20 tipi diversi di aminoacidi, ognuno con una propria catena laterale, la quale può essere apolare, carica, più o meno reattiva e così via…
La forma e la struttura di ogni proteina è caratterizzata dalle proprietà delle catene laterali (o gruppi R), in associazione alle limitazioni di movimento dovute al legame peptidico, alle proprietà degli altri legami interni agli aminoacidi e alle interazioni deboli tra i diversi domini della catena.
Il legame peptidico si forma quando l’atomo di carbonio del gruppo carbossilico di un aminoacido si lega covalentemente con l’atomo di azoto del gruppo amminico di un secondo aminoacido, con eliminazione di una molecola d’acqua. Il legame peptidico tra due aminoacidi adiacenti è il solo legame covalente all’interno della catena proteica e possiede un alto grado di stabilizzazione per risonanza.
Il singolo legame C-N infatti si comporta per il 40% come un doppio legame, esso è perciò rigido e non può ruotare liberamente, limitando notevolmente i vari tipi di conformazioni proteiche possibili.
Vi sono tuttavia altri due legami interni agli aminoacidi che invece possono ruotare liberamente, essi sono: il legame C-C di un aminoacido, il quale forma un angolo di rotazione detto “psi” e e il legame N-C di un altro aminoacido, con angolo di rotazione “phi”.
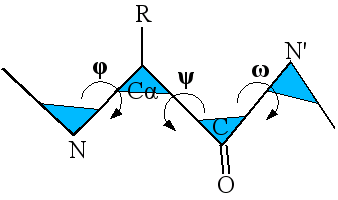
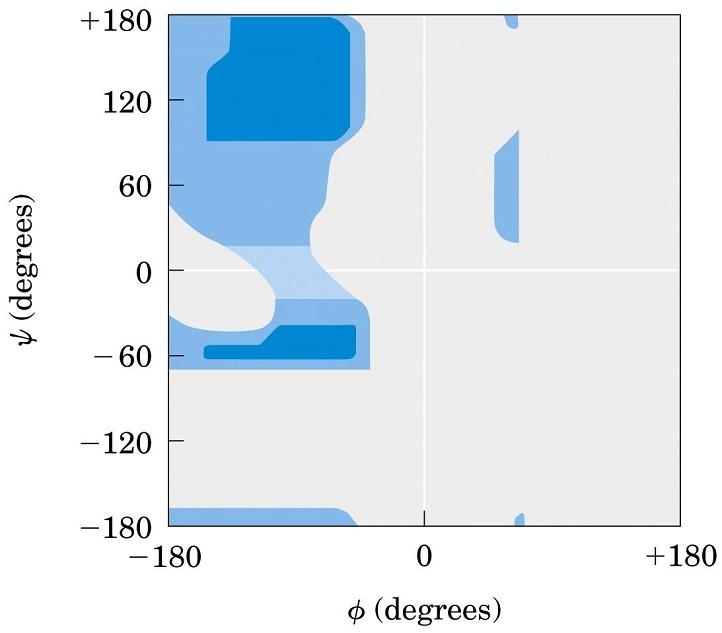
La conformazione degli atomi di una catena polipeptidica è perciò determinata dalla coppia di angoli psi e phi di ciascun aminoacido, i quali possono ruotare fino a quando le rispettive catene laterali degli aminoacidi non entrano in collisione tra loro.
Per prevedere gli angoli di legame, si utilizza il grafico di Ramachandran (vedi figura) in cui ciascun punto rappresenta una coppia osservata di angoli psi e phi di una proteina.
Questa libertà di conformazione permette così alla catena polipeptidica di ripiegarsi fino a formare notevoli strutture regolari.
Oltre alle proprietà specifiche dei legami intraaminoacidici, vi sono un insieme di forze deboli (non covalenti) che guidano il processo di ripiegamento della proteina nella sua conformazione nativa; questi legami sono: legami H, legami ionici, attrazioni di Van der Waals…
La stabilità complessiva della struttura dipende dalla somma delle suddette foze agenti.
Durante il processo di ripiegamento, detto “folding proteico”, la driving force che guida l’intero processo è rappresentato dalle proprietà idrofobiche delle catene laterali di alcuni aminoacidi che in soluzione tendono spontaneamente a disporsi lontano dalla fase acquosa, per ridurre al minimo l’energia totale del sistema.
Si ha così che i gruppi idrofobici degli aminoacidi come fenilalanina, leucina, valina e triptofano tendono a raggrupparsi internamente alla struttura proteica, mentre gli altri aminoacidi polari come arginino glicina e istidina presenti nella catena tendono a porsi sulla superficie della struttura, a diretto contatto con l’acqua (o altri solventi polari) dove stabiliscono legami idrogeno.
In termini energetici le proteine tendono sempre a disporsi in conformazioni con livelli di energia libera minimi (G°).
Nonostante l’informazione relativa alla conformazione proteica sia contenuta nella semplice sequenza di aminoacidi, allo stato della conoscenza attuale non si è ancora in grado di prevedere con assoluta certezza le precise conformazioni tridimensionali delle proteine.