Gli enzimi oligomerici possono essere suddivisi in due categorie:
mono-oligomeri: costituiti da monomeri tutti identici fra loro
etero-oligomeri: costituiti da monomeri differenti.
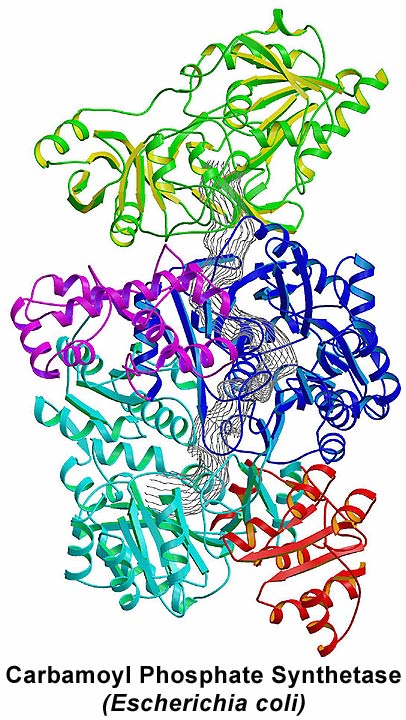
In questo secondo caso i monomeri possono avere funzioni diverse fra loro, in alcuni casi ciascun monomero può catalizzare una reazione diversa e spesso le reazioni sono strettamente regolate reciprocamente, come nel caso della carbamoil-fosfato-sintetasi di E. coli. Questo enzima è un α β eterodimero in grado di catalizzare tre reazioni differenti. http://www.chem.tamu.edu/rgroup/raushel/research/cps.html
Da: http://www.biochem.wisc.edu/rayment/lab/gallery_jpgs/cps.jpg
Nel caso di enzimi mono-oligomerici ogni subunità, essendo identica all’altra, dovrebbe avere lo stesso potere catalitico. Possono tuttavia verificarsi alcuni casi particolari:
-
può succedere che l’enzima ”funzioni” solo come dimero, associandosi e ”creando” grazie all’associazione l’unico sito attivo. La dissociazione in subunità rende inattivo l’enzima e questo è di fatto un meccanismo di regolazione.
-
in altri casi le subunità, seppur identiche, non hanno la stessa conformazione nello spazio e questo implica una maggiore funzionalità di una subunità rispetto all’altra che può anche essere totalmente inattiva. E’ questo il caso della Tirosil-tRna sintetasi di B. stearothermobilus. Pur avendo due subunità identiche tra loro, soltanto una catalizza la reazione di sintesi da tiroxina a tirosin-adenilato.
Per una esauriente trattazione di tali casi particolari, relativi sia ad enzimi mono-oligomerici che etro-oligomerici, allosterici e non, compresi i casi citati, si può consultare la Minireview:
“Interdomain interactions in oligomeric enzymes: creation of asymmetry in homo-oligomers and role in metabolite channeling between active centers of hetero-oligomers, di Natalya K. Nagradova (FEBS Letters 487 (2001) 327-332) “
GAPDH glyceraldehyde-3-phosphate dehydrogenase
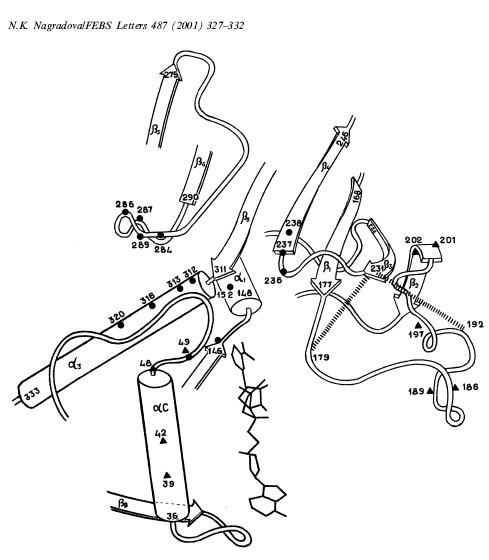
Frammenti della struttura tridimensionale di GAPDH.
Le linee in grassetto segnano gli elementi strutturali del dominio dove si lega il coenzima e quelle sottili del dominio catalitico. I circoli pieni indicano i residui di amino acidi che formano legami ad idrogeno tra i domini all’interno del monomero di E. coli. I triangoli pieni segnano i residui di aminoacidi che formano legami tra subunità vicine (contatti del tipo r). Sono mostrate anche due possibilità alterantive di legame tra Arg 231 e Thr 179 oppure Arg 231 e Asp 192.