La PCR (Polymerase Chain Reaction) è una tecnica ideata e messa a punto da Kary B. Mullis nel 1984, il quale ottenne per questo il premio Nobel per la chimica nel 1993. Questa tecnica permette di amplificare e ottenere copie, in numero esponenziale, di frammenti selezionati di acidi nucleici (DNA o RNA) in vitro.
I campi di applicazione della PCR sono enormi, grazie al fatto che qualsiasi materiale contenente DNA, come un fossile o tracce di sangue, può essere utilizzato da stampo per ottenere grandi quantità di copie degli acidi nucleici contenuti. Pertanto la PCR trova impiego nella diagnostica molecolare di malattie genetiche, del cancro, di malattie infettive, nell’archeologia, nel controllo di contaminazione di microrganismi negli alimenti, e soprattutto nel campo della ricerca scientifica dove ormai è ampiamente utilizzata.
Generalmente la PCR comprende una serie ciclica di tre fasi che si svolgono all’interno di un apposito strumento detto thermalcycler:
- denaturazione tramite calore di due filamenti di DNA;
- ibridazione per raffreddamento con primer in largo eccesso;
- sintesi di nuovo DNA per aggiunta della DNA polimerasi con conseguente allungamento dei primer.
In linea teorica ogni ciclo aumenterebbe la quantità di DNA in modo esponenziale, in realtà per avere una stima sufficientemente attendibile del numero di filamenti di DNA ottenuti si utilizza la seguente formula:
(1 + E)n
Dove n indica il numero di cicli effettuati ed E l’efficenza dell’amplificazione (in genere compresa tra 0,7 e 0,8).
La PCR si ottiene aggiungendo in una soluzione contenente il DNA stampo, grandi quantità di nucleotidi, delle sequenze primers (corte sequenze di DNA necessari alle attività delle polimerasi), e una DNA-polimerasi, ovvero un’enzima in grado di sintetizzare una nuova elica di DNA a partire da un’elica stampo.
Le DNA-polimerasi utilizzate attualmente sono tutte estratte da batteri che vivono in condizioni di temperatura estreme, in modo da essere termostabili. La “Taq” ad esempio, è in grado di lavorare ad alte temperature, fino anche a 92°C, temperatura alla quale avviene il processo di denaturazione del DNA. In passato questa DNA-polimerasi veniva estratta dal Thermophilus aquaticus, un batterio che vive nelle acque termali calde; attualmente invece viene prodotta in batteri opportunamente modificati per l’espressione e la sintesi dell’enzima.
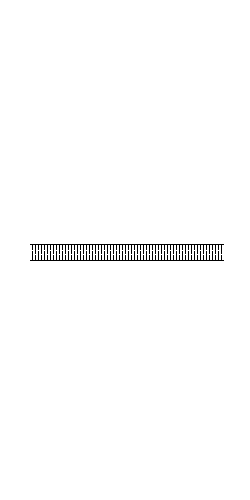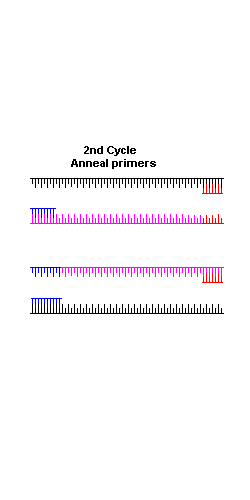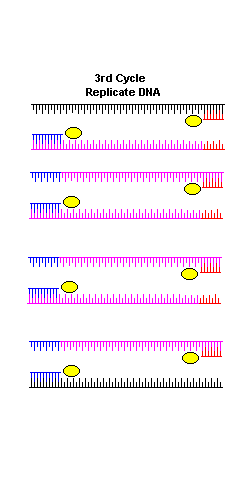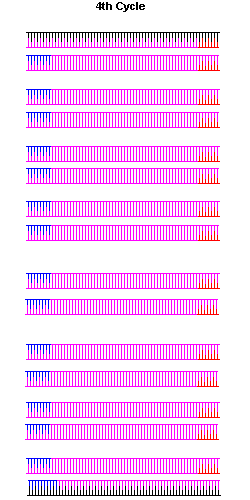
Nella prima fase il DNA estratto dalle cellule viene mescolato ad una miscela di reagenti che comprendono sali, nucleotidi, l’enzima Taq polimerasi e le sonde di innesco per l’enzima polimerasi, dette primer. Un primer non è altro che un piccolo frammento di DNA a singola elica, lungo circa 20 basi e complementare ad una sequenza di DNA che si vuole amplificare. Esso fornisce all’enzima Taq polimerasi un punto di innesco per la sintesi del nuovo filamento di DNA e ne servono due uno a monte ed uno a valle del tratto che si vuole amplificare. Ciascuno dei due primer si attacca ad un solo filamento. Nell’animazione sono i frammenti rossi e blu.
Il primer è aggiunto in grande eccesso al DNA. La provetta con la miscela di reagenti è posta nella macchina per PCR, capace di cicli successivi di riscaldamento e raffreddamento a temperature specifiche per ogni reazione. Le fasi della PCR si possono così riassumere:
-
denaturazione del DNA a temperature di 90-95°C
-
attacco del primer al sito bersaglio a temperature di 50-60°C, a seconda del protocollo specifico
-
fase di allungamento, a temperature di 72°C, ottimali per l’attività della Taq polimerasi, in cui l’enzima aggiunge nucleotidi all’estremità 3’ del primer
-
nuova denaturazione. Poi il ciclo da a) a c) si ripete per 20-30 volte, per finire con un ciclo di allungamento un po’ più lungo (anche fino a 1 minuto, o 5 minuti per l’amplificazione di loci micorsatelliti).
-
raffreddamento a 4°C per bloccare ogni rezione.
Attualmente esistono in commercio diversi tipi di PCR che si affiancano alla PCR tradizionale come la Real time PCR, la RT-PCR ( o PCR retro trascrizionale) per quantificare l’RNA in casi di scarsa abbondanza o ancora la LCR dove viene sfruttata l’azione di una ligasi termostabile.