La sequenziazione (o sequenziamento) è il processo che permette di ottenere la sequenza di una proteina o di una molecola di acido nucleico. La sequenza del DNA è l’insieme di nucleotidi letti nel giusto ordine. Nel DNA sono presenti 4 tipi di nucleotidi, contenenti una delle basi: A, G, C, T. A e G sono purine, C e T sono pirimidine. Se abbiamo un frammento di DNA di 300 nucleotidi e ne vogliamo determinare la sequenza, significa che vogliamo comprendere, nell’ordine esatto, quali sono i 300 nucleotidi. Alla fine del sequenziamento si ottiene un insieme di lettere, es. AAGTGTGTCTGTCAGTTCTAGAACC…, apparentemente prive di significato, in realtà a seconda dello studio che si sta effettuando avranno un proprio significato.
I metodi che si possono utilizzare per sequenziare il DNA sono:
1) Metodo di Maxam e Gilbert
2) Metodo di Sanger
3) Sequenziamento automatico del DNA in fluorescenza
Il metodo di Maxam e Gilbert prevede la marcatura delle estremità 3’ del DNA con 32P, il taglio del DNA e la separazione dei due filamenti identici marcati ad un’estremità. Si suddivide la miscela in quattro parti, ciascuna delle quali viene sottoposta ad un diverso trattamento chimico per metilare o rimuovere specificamente una o due basi. Si producono in questo modo frammenti marcati di dimensione specifica e tramite una elettroforesi è possibile determinare l’ordine dei nucleotidi e quindi la sequenza. Questo metodo veniva molto impiegato in passato, ora invece è poco utilizzato, perché non adatto al sequenziamento automatico e perché utilizza composti chimici dannosi alla salute.
Sanger è un biologo inglese vincitore di due premi Nobel: uno ottenuto per aver caratterizzato la struttura dell’insulina, l’altro ricevuto proprio per aver ideato un nuovo metodo di sequenziamento degli acidi nucleici.
Il metodo di Sanger viene anche definito metodo a terminazione di catena o metodo dei dideossinucleotidi.
Per il sequenziamento con il metodo di Sanger, si preparano in quattro provette le mix, costituite da:
-
Il campione di DNA che si vuole sequenziare a singolo filamento
-
la DNA polimerasi
-
un primer marcato con estremità 3’OH libera
-
i quattro tipi di deossinucleotidi trifosfato normali (dNTP) marcati con 32P o 35S
-
Infine in ogni provetta viene aggiunto uno specifico tipo di dideossinucleotidi trifosfato (ddNTP), in una provetta ddA, in un’altra ddG, in un’altra ddC ed in un’altra ddT, i ddNTP sono la chiave del successo di questa tecnica.
Si verifica che il primer complementa con il filamento stampo e viene esteso dalla DNA polimerasi, questa infatti aggiunge all’estremità libera del primer i nucleotidi complementari al filamento stampo, cioè i dNTP, invece di aggiungere un dNTP di tanto in tanto può accadere che aggiunge un ddNTP. Con l’incorporazione del ddNTP la sintesi del filamento complementare si ferma poiché i ddNTP non permettono la formazione del legame con il successivo nucleotide trifosfato. I dNTP hanno un 3’OH nello zucchero desossiribosio che consente l’aggiunta di nucleotidi, i ddNTP invece hanno un 3’-H che non consente l’aggiunta di ulteriori nucleotidi e per questo la sintesi del DNA termina.
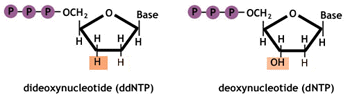
La stessa cosa accade per tutti i frammenti di ciascuna provetta ovvero si ha la sintesi del filamento complementare fino a che non viene incorporato un ddNTP. Si ottengono in questo modo frammenti di DNA di varia lunghezza, che differiscono da provetta a provetta per il tipo di ddNTP con il quale terminano. I campioni marcati contenuti nelle quattro mix vengono poi denaturati al calore e caricati in quattro pozzetti diversi, in un gel di poliacrilammide. L’elettroforesi viene realizzata a circa 70°C in presenza di urea in modo da impedire la rinaturazione del DNA. Con l’elettroforesi i frammenti migrano in base alla dimensione e quindi al termine della elettroforesi, mediante autoradiografia, è possibile risalire alla sequenza.
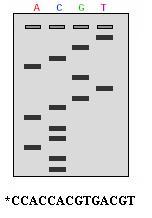
Esempio di gel ottenuto con il metodo di Sanger
La sequenza viene letta dal basso del gel verso l’alto. Nella prima corsia elettroforetica (A), si osservano 3 bande, questo significa che in quelle posizioni è stato incorporato un ddATP e quindi significa che in quelle 3 posizioni c’è una A nella sequenza. Si procede con lo stesso ragionamento per le altre 3 corsie elettroforetiche che sono indici di dove sono posizionati i nucleotidi C, T, G. In basso è riportata la sequenza del filamento.
Attualmente esistono metodi di sequenziamento automatico, la sintesi dei filamenti procede come già descritto prima, però con questa metodica vengono incorporati, per terminare la sequenza, ddNTP marcati con fluorocromi diversi. I prodotti di sintesi vengono fatti correre sulla stessa corsia di un gel denaturante, un laser eccita i fluorocromi, ciascuno dei quali emette una luce d’onda caratteristica e tramite un sistema automatizzato viene riportata su un personal computer la corretta sequenza nucleotidica del frammento analizzato. La procedura è completamente automatizzata ed in questo modo si ha un grosso risparmio in termini di tempo, poiché si possono analizzare più campioni simultaneamente.
Da visitare:
http://www.bioteach.ubc.ca/Bioinformatics/GenomeProjects/
http://www.dnalc.org/shockwave/cycseq.html
Fatto molto bene e molto chiaro!