La tecnica dell’RNAi deriva dal concetto generale che un RNA, in presenza di una catena di RNA complementare, formi con questo un doppio filamento molto stabile. L’RNA a doppio filamento non viene tradotto e quindi viene abolita la sintesi proteica. La presenza di questa molecola di RNA a doppio filamento della cellula, porta a quell’effetto chiamato di interferenza dell’RNA (RNAi).
L’effetto RNA interference si è osservato per la prima volta in alcune piante di petunia nelle quali era stato transinfettato un gene soprannumerario con un promotore della trascrizione indipendente, allo scopo di intensificarne il colore. Il gene codificava per una proteina uguale a quella già presente nell’organismo. Il risultato fu alquanto strano, dal momento che le petunie ottenute non presentavano un colore più intenso ma risultavano screziate e qualche individuo era completamente bianco. Questo fenomeno va sotto il nome di silenziamento genico post trascrizionale (PTGS).
La spiegazione a questo fenomeno è stata trovata solo negli anni ’90 quando si scoprì che un RNA a doppio filamento promuoveva la degradazione dell’RNA messaggero. L’effetto RNAi è probabilmente un meccanismo molto antico di difesa contro le infezioni da virus a RNA. Probabilmente l’RNA a doppio filamento ricorda alla cellula un genoma virale o un trasposone (trasposoni = elementi trasponibili a vita ’indipendente’ che sono presenti talvolta nel genoma e che si autoreplicano e si diffondono sia orizzontalmente che verticalmente).
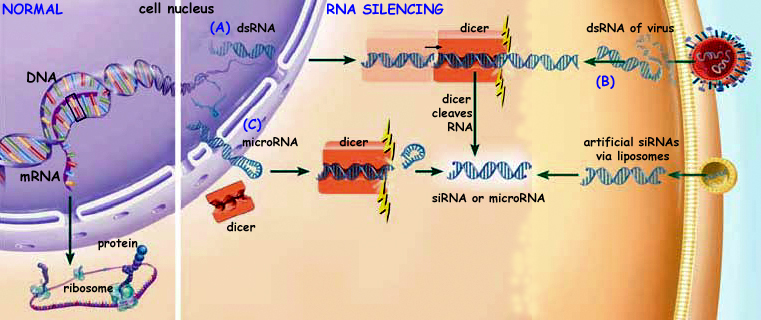
Reazioni della cellula all’ingresso di un dsRNA (double stranded = a doppia elica) di origine virale.
Quindi l’organismo risponde producendo o attivando RNA-endonucleasi che tagliano l’RNA ds in tratti di 21-23 nucleotidi. Sembra che in questo primo passo sia coinvolto il gene Dicer, appartenente alla famiglia delle RNase di classe III. Ad un certo stadio inoltre questo segnale di allarme viene amplificato dalla duplicazione dell’RNA estraneo attraverso una RNA polimerasi RNA-dipendente. I frammenti di 21 nucleotidi vengono ulteriormente degradati a duplex di 19-21 nucleotidi, che presentano estremità sporgenti costituite da due nucleotidi. Questi frammenti di RNA prodotti sono a singolo filamento e possono legarsi all’RNA ospite impedendone la traduzione.
I geni coinvolti nel processo sono ancora poco conosciuti, soprattutto nei mammiferi, per i quali è stata al momento trovata una implicazione per un membro della famiglia RDE-1. Alcuni dei geni coinvolti nel processo di RNAi sembrano attivati anche nel caso di distruzione dell’mRNA mediata da sequenze non senso, un meccanismo che permette alle cellule di controllare gli mRNA appena trascritti e di impedire la produzione di proteine difettive. Sembrano essere legati all’effetto RNAi anche tre geni della famiglia SMG.
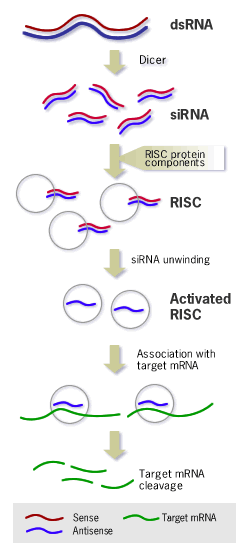
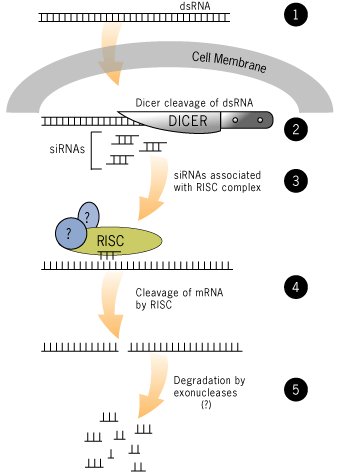
Due schemi esplicativi del meccanismo di azione del RNA a doppio filamento nelle cellule.
Il metodo dell’RNAi per il silenziamento dei geni è stato sviluppato in Cenorhabditis elegans nel 1998 (un nematode):
I piccoli RNAi (siRNA) artificiali utilizzati sono RNA a doppio filamento di 19-21 nucleotidi con estremità che presentano 2 nucleotidi simmetrici sporgenti, preferibilmente due timidine. I siRNA vengono introdotti nelle cellule, dove si associano con proteine specifiche formando complessi ribonucleoproteici che setacciano gli mRNA contenuti nel citoplasma per degradare in maniera selettiva solo quello che contenga la sequenza corrispondente.
La tecnica dell’RNA interference (RNAi) è stata usata con successo in molteplici organismi come il moscerino della frutta, in funghi e piante, nel nematode Cenorhabditis elegans. Ultimamente è stato scoperto che l’effetto RNAi viene utilizzato normalmente da molti organismi. In C. elegans, ad esempio esistono dei small temporal RNA (stRNA) che ne regolano lo sviluppo attraverso una repressione transitoria sdi specifici trascritti, questi stRNA vengono prodotti dalla proteina DICER a partire da precursori di circa 70 nucleotidi. Parimenti, sia in Drosophila che nelle cellule HeLa esistono RNA endogeni che causano PTGS e che vengono prodotti dal taglio di trascritti più lunghi. Nelle cellule di mammifero tuttavia la tecnica classica non ha mai dato risultati soddisfacenti. Inizialmente problemi e insuccessi si sono verificati in generale con i vertebrati, solo in zebrafish nel 1999 si era riusciti a ottenere un silenziamento, se pur solo per alcuni geni. La motivazione del fallimento dell’RNAi nei mammiferi è dovuto al fatto che la presenza di RNA a doppio filamento, tipico sintomo di infezione virale, causa una risposta mediata da interferone che blocca la traduzione in maniera aspecifica e porta ad apoptosi della cellula. Negli organismi nei queli l’RNAi funziona invece, l’infezione virale viene combattuta degradando i trascritti virali e quindi impedendone la moltiplicazione.
Esistono due vie di azione dei trascritti lunghi a doppio filamento (dsRNA) nei mammiferi. La prima via prevede che i dsRNA attivino la PKR che fosforila e inattiva il fattore di iniziazione della traduzione. Il secondo pattern coinvolge invece l’attivazione della RNase I che degrada gli mRNA cellulari in maniera indiscriminata. È stato recentemente scoperto che la via delle PKR non viene attivata da dsRNA di lunghezza inferiore ai 30 nucleotidi, chiamati small interference RNA (siRNA). L’utilizzo di siRNA nelle celllule di mammifero permette il silenziamento genico specifico e il meccanismo è così preciso che una sola mutazione dei 21 nucleotidi del siRNA rispetto al mRNA bersaglio annulla quasi completamente il silenziamento. Nei mammiferi resta tuttavia il problema di disegnare i siRNA corretti, dal momento che non tutti sembrano funzionare correttamente.
Per questo motivo sono stati creati vettori in grado di far esprimere small hairpin RNA (shRNA) che vengono processati in vivo a dare molecole di siRNA. I vettori contengono la sequenza del shRNA tra un promotore per la polimerasi III e un sito di terminazione. La scelta della polimerasi III non è casuale, dal momento che questo enzima non aggiunge le code di poly-A permettendo quindi al shRNA di ripiegarsi in una struttura a forcina. Un secondo metodo prevede invece cha il vettore contenga due promotori della polimerasi III in grado di creare un filamento antisenso e uno senso, ottenendo direttamente un siRNA di diversi RNAi utilizzando vettori.
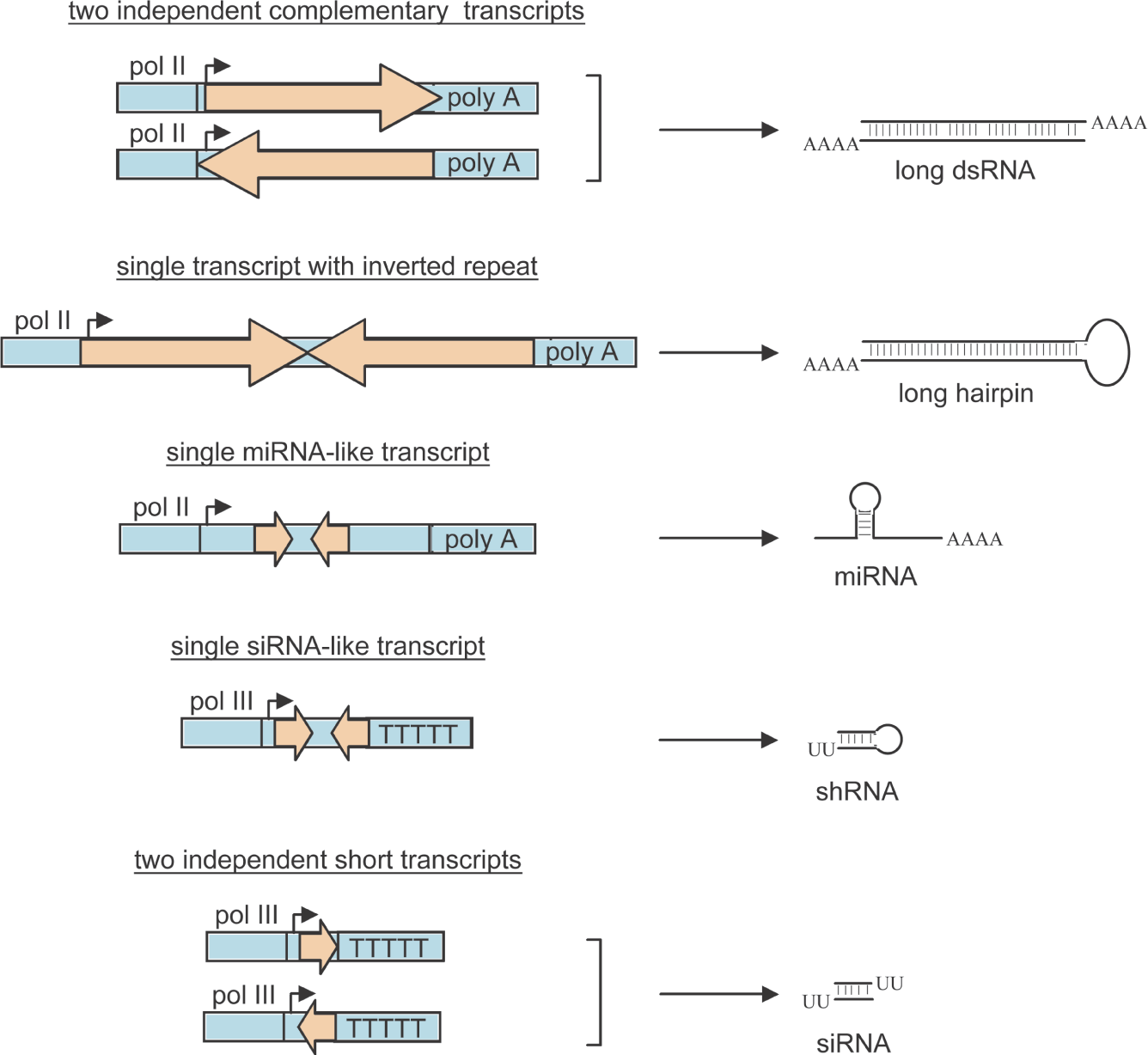 Produzione
Produzione
Entrambi questi tipi di RNA si legano alla sequenza dell’RNA messaggero (mRNA), a loro complementare, presente nella cellula, rendendone impossibile la traduzione e causandone la degradazione. Il risultato è l’annnullamento della sintesi di una determinata proteina bersaglio.
Il metodo vector-based per l’RNA interference rende possibile lo spegnimento di geni in maniera molto specifica nella cellula differenziata e il suo effetto è persistente, al contrario dei primi metodi per l’RNAi utilizzati nei mammiferi.
Qui di seguito si trovano alcuni link con spiegazioni più approfondite:
hal9000.cisi.unito.it/wf/DIPARTIMEN/ Genetica,_/Attivit–D1/Biologia-m/RNAi.ppt
case 12