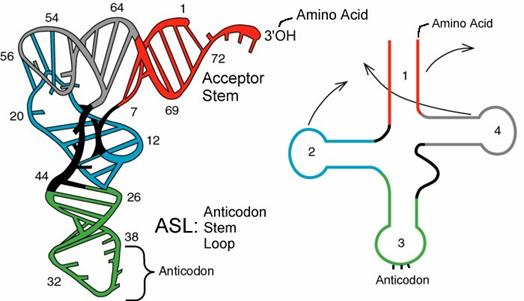
Il tRNA o RNA di trasporto, è un particolare tipo di RNA che funge da tramite tra l’RNA messaggero e la proteina che esso codifica. Esistono numerosi tipi di RNA di trasporto, teoricamente uno per ogni codone codificante presente sull’mRNA, e presentano tutti strutture secondarie e terziarie comuni. La struttura secondaria del tRNA viene comunemente rappresentata come un quadrifoglio, in cui l’appaiamento complementare delle basi forma steli che separano anse a singolo filamento.
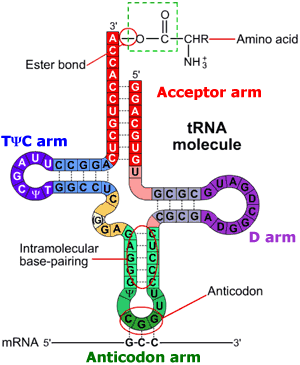
Fig 1. Diagramma della struttura secondaria di un tRNA classico. Sono indicate le regioni principali e le loro interazioni. Da www3.interscience.wiley.com.
I bracci del tRNA, cioè l’insieme di un’ansa con il proprio stelo, comprendono nella loro sequenza basi insolite che sono generate dalla modificazione delle 4 basi standard dopo la sintesi della catena polinucleotidica. I quattro bracci principali prendono il nome dalla loro struttura o funzione.
Braccio accettore: è unito ad un aa
Braccio TψC: presenta i le tre basi Timida, Pseudouridina, Citosina. (ψ sta per pseudouridina)
Braccio dell’anticodone: contiene la tripletta dell’anticodone al centro dell’ansa.
Braccio D: contiene la base diidrouridina
Braccio extra: sta tra il braccio TψC e quello dell’anticodone ed è composto da 3 a 21 basi.
La struttura secondaria di ciascun tRNA si ripiega in una struttura terziaria compatta a forma di L: l’aa si trova all’estremità di un braccio e l’ansa dell’anticodone forma l’altra estremità.
Fig 2. Confronto tra struttura secondaria e terzaria del tRNA. www2.kenyon.edu/Depts/BioEllipse/courses/biol63/ribo/ribo.html
Poiché l’anticodone è complementare al codone, è la prima base dell’anticodone scritto in direzione 5’-3’ che si appaia con la terza base della sequenza del codone.
Codone 5’ A C G 3’
Anticodone 3’ U G C 5’
L’interazione tra l’anticodone e il codone avviene per appaiamento delle basi, ma secondo regole che estendono l’appaiamento oltre le solite coppie G-C e A-U. In quasi tutti i casi o la terza base è irrilevante o viene fatta una distinzione soltanto fra purine e pirimidine. Esistono solo tre casi in cui un significato unico è conferito dalla presenza di una base particolare nella terza posizione. Questa particolarità permette a un tRNA di riconoscere più di un codone, il che significa che la base nella prima posizione dell’anticodone deve essere capace di appaiarsi a basi alternative nella terza posizione corrispondente del codone.
L’eccezione nell’appaiamento delle due triplette, codone e anticodone, viene spiegata attraverso l’ipotesi del tentennamento. Secondo questa teoria, l’appaiamento fra il codone e l’anticodone a livello delle prime due posizioni del codone segue le solite regole, mentre a livello della terza posizione si verifica un tentennamento eccezionale, permesso dalla conformazione dell’ansa dell’anticodone del tRNA.
Il tRNA è l’unico fra gli acidi nucleici a contenere basi insolite, più di 50 tipi differenti, prodotte per modificazioni di una delle quattro basi dopo l’incorporazione nella catena poliribonucleotidica. Nei tRNA si osserva una gamma di modificazioni che vanno dalla semplice metilazione ad un’estesa ristrutturazione dell’anello purinico. Esempi di queste basi modificate sono la diidrouridina (D), generata per saturazione di un doppio legame che cambia la struttura dell’anello, la pseudouridina (ψ), nella quale sono scambiate le posizioni degli atomi di N e di C e la tiouridina, che possiede uno zolfo a sostituire un ossigeno.
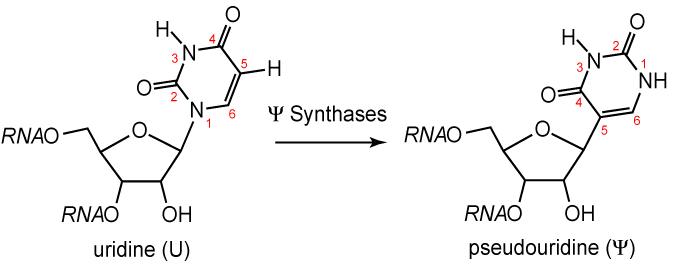
Fig 3. Un esempio di base azotata modificata, la pseudouridina. da http://en.wikipedia.org/wiki/Pseudouridine
L’importanza di queste modificazioni nelle basi azotate è particolarmente evidente a livello dell’anticodone, nel quale la loro presenza permette schemi di appaiamento non classici: l’inositolo, ad esempio, si può appaiare con U,C ed A. Inoltre, alcune modificazioni creano letture preferenziali di alcuni codoni rispetto ad altri.
Il processo della sintesi proteica, alla cui base sta il riconoscimento codone/anticodone e qundi la traduzione del codice genetico in sequenza amminoacidica, avviene a livello di un organello chiamato ribosoma. Il ribosoma presenta due siti di legame per i tRNA, un sito P e un sito A e gioca un ruolo importante nella specificità dell’interazione codone/anticodone. La fedeltà della sintesi proteica è determinata infatti dall’interazione di un codone dell’mRNA con l’anticodone del tRNA corretto a livello del sito A del ribosoma, il quale è in grado di riconoscere gli accoppiamenti codone-anticodone corretti da quelli errati. La specificità dell’accoppiamento codone-anticodone è di per sé abbastanza bassa ed è il ribosoma che assicura un’elevata fedeltà di traduzione attraverso un meccanismo di discriminazione cinetica che accoppia il riconoscimento della coppia codone-anticodone da parte del ribosoma con l’idrolisi del GTP da parte del fattore di elongazione Tu.
La struttura canonica dell’ansa dell’anticodone è essenziale per l’interazione sia con il sito A che con il sito P ed è evolutivamente conservata. L’ansa dell’anticodone è caratterizzata dalla presenza di un certo numero di nucleotidi conservati o semiconservati che formano un’estesa firma strutturale. All’estremità 5’ dell’ansa, si trova una base pirimidinica in posizione 32, seguita da un U invariante alla posizione 33. In posizione 34 si trovano diversi nucleotidi modificati che si accoppiano con il nucleotide in posizione 3 del codone, mentre i nucleotidi 35 e 36, che interagiscono con le posizioni 1 e 2 rispettivamente del codone, mostrano modificazioni solo limitatamente. Le basi in posizione 37 e 38 sono frequentemente ipermodificate e studi su sistemi modello hanno indicato che le modificazioni della base in posizione 37 stabilizzano le interazioni tra mRNA e tRNA.
Probabilmente la funzione più importante delle modificazioni del tRNA, inclusa quella alla posizione 3, è l’effetto sull’accuratezza della lettura del codone al sito A.
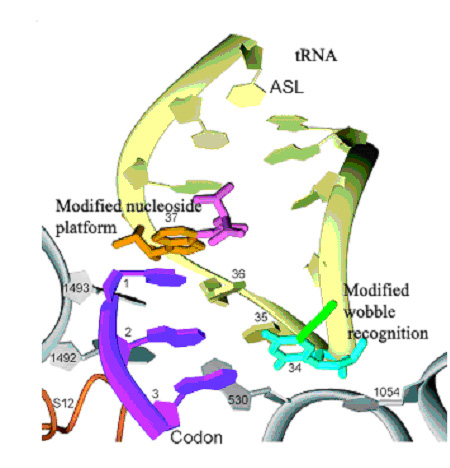
Fig 4. Schema della zona di interazione tra codone (viola) e anticodone (giallo) ottenuto tramite elaborazione di dati atomici. Da http://www.ncsu.edu/news/press_releases/04_12/307.htm. I due nucleotidi modificati in posizione 37 e 34 interagiscono in maniera fisica con il codone, stabilizzandone l’accoppiamento con l’anticodone corretto.
Un aspetto notevole della biosintesi delle proteine è la sua precisione. Stime della frequenza di errore nella traduzione suggeriscono che nella proteina venga inserito solo un amminoacido errato ogni 300 incorporati. Questa precisione non è spiegabile basandosi soltanto su quello che si sa circa il meccanismo di selezione dell’aminoacil-tRNA. Infatti, misure di affinità dei trinucleotidi per le loro sequenze complementari nell’anticodone delle molecole di tRNA indicano che tale interazione è abbastanza debole e che può essere caratterizzata da una costante di equilibrio dell’ordine del 103 M-1 a 0°. Inoltre un dato trinucleotide può interagire con la sua molecola di tRNA accoppiata con una costante di affinità che è solo 10 volte maggiore di quella per una tripletta non complementare con lo stesso tRNA. Tale osservazione suggerisce che una semplice interazione tripletta-tripletta all’equilibrio non può essere la sola motivazione per la fedeltà della selezione di tRNA.
Il legame delle molecole di tRNA sul ribosoma è accompagnato da riarrangiamenti strutturali, ad esempio il legame dell’aminoacil-tRNA alla subunità ribosomiale minore è accompagnato dall’esposizione di uno o più siti che possono legare il tetranucleotide CGAA. Questa sequenza è complementrare alla sequenza TψC presente sul tRNA. L’aspetto più interessante di questo particolare sistema di riconoscimento è che il corretto accoppiamento tra le due triplette in gioco aumenti l’accessibilità del sito di legame CGAA.
E’ probabile che la struttura del tRNA si sia evoluta in maniera tale che quando l’anticodone interagisce con il suo codone accoppiato sul ribosoma, la piega dell’ansa dell’anticodone viene alterata. Tale transizione conformazionale corrisponde al primo stadio nel processo di selezione del tRNA. Il riarrangiamento dello scheletro dell’ansa dell’anticodone, quindi, inizia una serie di cambiamenti conformazionali nel tRNA che portano allo smascheramento dei siti di legame per il ribosoma in altre posizioni del tRNA, come l’ansa D e l’ansa TphiC. Dal momento che questi cambiamenti conformazionali sono dipendenti dall’nterazione corretta codone-anticodone, essi contribuiranno alla specificità dipendente dal codone della reazione di legame del tRNA. Quindi, il rafforzamento dell’interazione del tRNA complementare con il ribosoma è ottenuta attraverso siti di legame non specifici che vengono messi in gioco dai cambiamenti conformazionali dipendenti dal codone del tRNA.
Questo sito presenta alcune animazioni riguardanti la traduzione e i fattori in essa attivi.
Link di approfondimento sul ruolo delle basi modificate nell’interazione codone-anticodone